Wie kommt es, dass jede Zelle des menschlichen Körpers die gleiche Erbinformation besitzt – und doch unterschiedliche Funktionen erfüllt? Warum können Umwelteinflüsse wie Feinstaub auf Dauer krank machen? Und Sport und eine ausgewogene Ernährung gesund? All das und mehr in diesem Beitrag.
„Die Epigenetik verändert nicht den genetischen Code, sondern die Art und Weise, wie er gelesen wird. Völlig normale Gene können zu Krebs oder Tod führen. Umgekehrt werden mutierte Gene in der richtigen Umgebung nicht exprimiert. Gene sind mit Bauplänen vergleichbar; die Epigenetik ist der Bauunternehmer. Sie verändern den Aufbau, die Struktur.“
Bruce Lipton
Die wissenschaftliche Disziplin der Epigenetik beschäftigt sich mit Molekülen und Mechanismen, die alternative Aktivitätszustände von Genen aufrechterhalten, ohne die DNA-Sequenz selbst zu verändern 1. Einfacher gesagt: es geht um biologische Schalter, die Gene langfristig an- oder abschalten nach Bedarf.
Im nicht streng wissenschaftlichen Sprachgebrauch werden häufig auch nicht vererbbare Mechanismen dazu gezählt, oder jene, die zeitlich begrenzt sind. Doch selbst in der Wissenschaft ist die Definition von Epigenetik ein Streitpunkt 2. Denn die Bedeutung des Wortes hat sich seit der Prägung durch den Genetiker Conrad Waddington in den 1940ern stetig gewandelt 3.
Inhaltsverzeichnis
- 1 Gene ausschalten – wozu?
- 2 Histone – mehr als nur Verpackung
- 3 DNA-Methylierung: weniger ist mehr
- 4 Nicht codierende RNAs – kleine und große Helferlein
- 5 Generationsübergreifende Vererbung – auch beim Menschen?
- 6 Traumata und epigenetische Auswirkungen auf Kinder
- 7 Gedächtnis wäre ohne das Epigenom nicht möglich
- 8 Die molekulare Uhr – genetische Anzeichen für Alterung
- 9 Verhalten und Umwelt verändern die Gene
- 10 Weiterführende Links
- 11 Quellen
Gene ausschalten – wozu?
Regulation der Gene ist notwendig, um Energie zu sparen. Wer braucht schon einen Hammer zum Bohren? Außerdem ermöglichen verschiedene Lesearten desselben genetischen Bauplans, dass Zellen zu 220 unterschiedlichen Geweben werden können und ihre spezifische Funktion ausüben (Abbildung 1). Wir verlassen uns darauf, dass neue Zellen in unserem Herz weiterhin zu Herzzellen werden, und nicht gar zu Leberzellen. Genau das wird durch epigenetische Schalter ermöglicht.
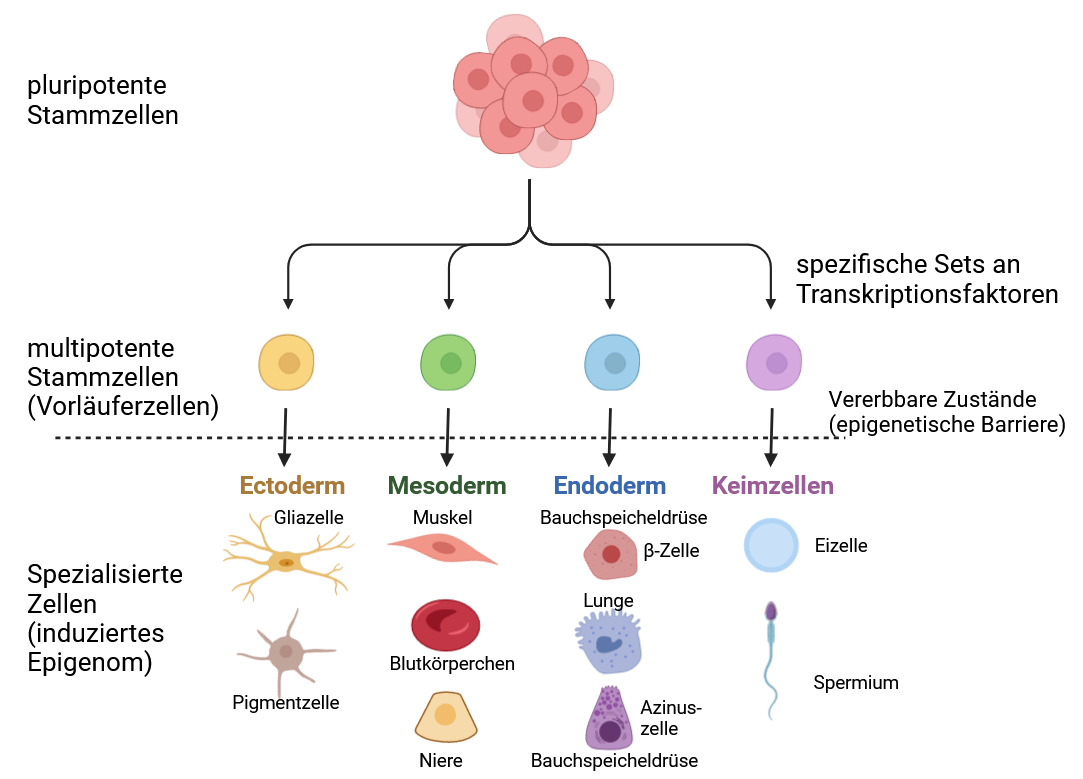
Aber auch Flexibilität spielt eine wichtige Rolle. Epigenetische Vorgänge sind immer reversibel, um auf Veränderungen in der Umwelt oder Stress reagieren zu können.
Nicht nur die eigenen Essgewohnheiten, sondern auch jene der Eltern vor unserer Zeugung tragen dazu bei, ob man ein erhöhtes Risiko für Gewichtsprobleme oder Krankheiten haben kann 4,5. Somit startet jeder Mensch nicht nur mit unterschiedlichen sozio-ökonomischen oder kulturellen Gegebenheiten ins Leben. Auch genetisch können wir vorbelastet sein. Doch ist man seinem Schicksal keineswegs nur ausgeliefert. Positive wie negative Gewohnheiten der Lebensführung beeinflussen unseren epigenetischen Code nachhaltig. Wie funktioniert das?
Histone – mehr als nur Verpackung
Wenn man sich an den Vergleich der DNA (Desoxyribonukleinsäure) mit einer Filmrolle aus dem letzten Beitrag über mRNA-Impfstoffe (Backlink) erinnert, dann besteht die Rolle aus Histonen, während das Filmband die DNA repräsentiert. Histone sind also diejenigen Proteine, auf denen die DNA im Zellkern aufgewickelt vorliegt (Abbildung 2). Zum einen, um Platz zu sparen. Komplett entknotet wäre jede DNA pro Zelle nämlich 2 Meter lang. Zum anderen zur Genregulierung.
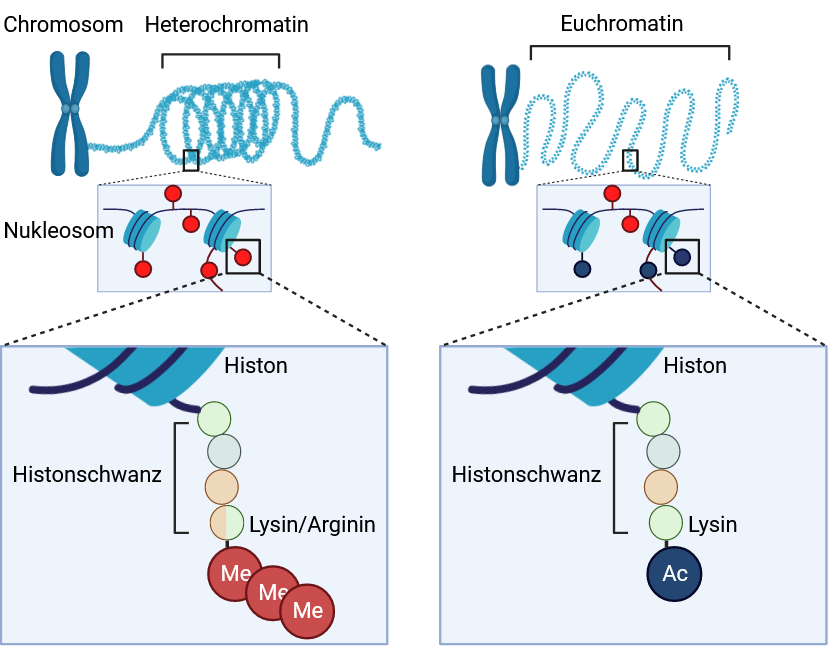
Je nachdem, wie dicht gepackt diese Histone sind, können gewisse Gene exprimiert werden, also erst in mRNA und dann funktionstüchtige Proteine umgewandelt, oder bleiben räumlich unerreichbar für das Ablese-Enzym DNA-Polymerase. Wie weit die als Nukleosom bezeichneten Filmrollen geöffnet vorliegen, kann durch Bindung von verschiedenen chemischen Molekülen beeinflusst werden 6. Dabei sind besonders die aus dem Nukleosom hervorstehenden losen Enden, oder Histonschwänze, empfänglich für epigenetische Marker.
Bindet eine negative Acetylgruppe ein herausragendes Lysin (Aminosäure) des Histons, wird die statische Interaktion des Lysins mit der negativen Ladung der DNA räumlich begrenzt aufgehoben. Dadurch lockert sich die übergeordnete Struktur des Chromatins. Genug, damit die DNA-Polymerase binden, und die entsprechende Sequenz ablesen und in mRNA übersetzen kann. Diese aktive Formation wird Euchromatin genannt.
Binden hingegen bis zu drei Methylgruppen auf einmal am Histonschwanz, nimmt die Dichte des Chromatins vereinfacht gesagt zu. Dadurch wird die DNA enger aufgerollt und für die Transkription „verschlossen“ oder stumm geschaltet. Diesen dichten Packungszustand nennt man Heterochromatin. In der Realität können Methylierungen sowohl anregend als auch reduzierend auf die Genaktivität wirken, je nach Muster und Bindungsstelle. Außerdem können weitere chemische Moleküle binden, zum Beispiel Phosphat oder Ubiquitin. Deren Wirkung ist jedoch noch weitestgehend unerforscht.
Ob Histon-Modifikation aber wirklich zur Epigenetik gehört, war unter Wissenschaftlern lange umstritten 7,8, da Epigenetik per definitionem vererblich, reversibel und selbsterhaltend ist 8. Anders gesagt: ein Gedächtniseffekt muss gegeben sein, so dass ein Gen auf Dauer aktiv oder stumm bleibt, auch nachdem das ursprüngliche Signal nicht mehr da ist. Dieses Gedächtnis muss in irgendeiner Art nach der Zellteilung bestehen bleiben. Besonders die Vererbarkeit und Selbsterhaltung wurden angezweifelt 7.
Denn sobald epigenetische Marker nicht mehr an Histone gebunden sind, verfallen sie in ihren Grundzustand zurück. Inzwischen sind mehrere Systeme bekannt, die dafür sorgen, dass Chromatin verändernde Proteine weiterhin ihren Job machen und ihrem Gedächtnis auf die Sprünge helfen 9,10.
In den letzten Jahren versteht man immer besser, wie die epigenetischen Marker der Histone während der Zellteilung (Mitose) erhalten bleiben 1,11,12.
Doch inwieweit Histon-Modifikationen wirklich an Nachkommen vererbt werden, und wenn ja, durch welchen Mechanismus, ist noch nicht abschließend geklärt. Eine Hypothese lautet, dass die (extrem) wenigen epigenetischen Marker, die vererbt werden 13, ausreichen sollen um die Chromatinstruktur wiederherstellen 14.
Jedoch ist die 3D-Struktur des Genoms und die einzelne Positionierung der Nukleosomen darin keinesfalls Zufallsprodukt und ebenso bedeutsam wie die Packungsdichte. Diese räumliche Anordnung kann vererbt werden und agiert außerdem als Bindeglied zum nächsten Mechanismus 15.
DNA-Methylierung: weniger ist mehr
Der zweite epigenetische Faktor ist die direkte Methylierung der DNA: die Bindung eines Methylmoleküls an ein Cytosin, welches von einem Guanin (zwei der vier Basen bzw. Bausteine der DNA) benachbart wird. Im Fachjargon heißt die Konstellation CpG-Dinukleotid oder -Stelle. Eigentlich sind CpG Dinukleotide in Säugergenomen selten, doch sie liegen gehäuft in etwa 1000 Basenpaaren langen CpG-Inseln vor. Durch Bindung des Methyls entsteht 5-Methylcytosin (5mc), welches dazu neigt, zu Thymin (einer weiteren Base) zu mutieren 16. Trotz dieser Instabilität sind circa 70-80% aller CpG Dinukleotide in Säugergenomen methyliert, was auf ihre Wichtigkeit deutet 17.
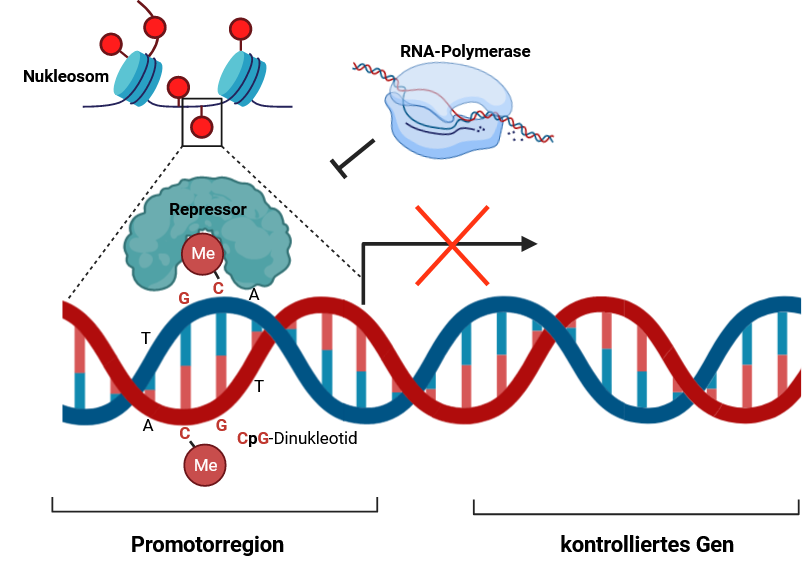
An diesen Markierungen können sich gewisse Proteine, sogenannte Transkriptionsfaktoren, mit Methyl CpG Bindedomänen orientieren und binden 16. Diese Transkriptionsfaktoren bestimmen dann darüber, ob das Gen abgelesen wird oder stumm bleibt (Abbildung 3).
Vereinfacht gesagt wird Methylierung normalerweise mit der Stummschaltung von Genen, und Demethylierung, also das Entfernen des Moleküls, mit Genaktivierung verbunden 16,18.
Das spielt zum Beispiel bei der Zelldifferenzierung (Abbildung 1) oder der Inaktivierung des redundanten X-Chromosoms in Frauen eine Rolle. Ebenfalls gibt es den Effekt der genetischen Prägung (engl. Imprinting), das bedeutet der geschlechter-abhängigen Methylierung mancher Gene. In etwa 220 menschlichen Genen sind so entweder die väterlichen oder mütterlichen Allele (Genvarianten) aktiv, aber nie beide 19. Normalerweise werden die Methylierungsmuster in einer frühen Phase der Keimzellausbildung in Embryos gelöscht, doch manche Regionen widerstehen dieser Umprogrammierung 20.
Andererseits ist die unkontrollierte Demethylierung, meist als Folge von mutierten Kontrollmechanismen, ein Grundbestandteil aller Krebszellen. Diese wuchern folglich unkontrolliert, weil zu viele (Wachstums-) Gene gleichzeitig aktiviert sind 1.
Die symmetrische DNA-Methylierung wird bei der Vervielfältigung der DNA während der Zellteilung (Mitose) kopiert. Somit kann der Aktivitätszustand einzelner Gene an die nächste Zelle weitergegeben werden.
Bei Bienen bestimmt das Methylierungsmuster im „Gehirn“ darüber, ob eine Arbeiterin oder Königin aus einer Larve entsteht 21. Die Nachkommen, die zur Königin heranwachsen sollen, werden in besondere, größere Waben gelegt und bekommen dort das Gelée Royal. In dieser Nahrung der Champions ist unter anderem auch Phenylbutyrat enthalten, ein bekannter Histon-Deacetylase-Inhibitor 22. Wie wir gelernt haben, wirken Acetylreste an Histonen auflockernd auf das Chromatin. Wird die Acetyl-entfernende Deacetylase gehemmt (inhibiert), kann die betroffene Genregion also weiterhin transkribiert werden.
Nicht codierende RNAs – kleine und große Helferlein
Der dritte Faktor epigenetischer Regulation sind nicht-codierende RNAs (non-coding; ncRNAs) 23. Im Gegensatz zu messenger RNAs (mRNA), werden ncRNAs nicht in Proteine übersetzt. Prominente Beispiele sind die Ribosomen, welche an der Übersetzung von mRNA zu Proteinen beteiligt sind. Doch regulatorische Elemente machen wohl nur 3% des Genoms aus.
Allgemein codieren nur etwas 1-2% des menschlichen Genoms für Proteine (ca. 22.000), während der Großteil als „Junk“ DNA, also Schrott DNA, bezeichnet wird. Viele Abschnitte stammen ursprünglich aus sich selbst replizierenden DNA-Sequenzen (Transposons) oder Viren, üben aber keine offen ersichtliche Funktion aus. Man vermutet, dass diese sich wiederholenden Sequenzen zur 3D Struktur des Genoms beitragen, indem sie auch entferne Abschnitte zusammenlagern und so die Genexpression beeinflussen.
2012 veröffentlichte ein Forscherteam Ergebnisse des ENCODE Projektes (Encyclopedia of DNA elements), wonach bis zu 94% des Genoms in RNA übersetzt werden soll 24.
Für die Epigenetik bedeutsame ncRNAs sind 1) die micro– oder miRNA, 2) die small interfering– oder siRNA, 3) die piRNA (Piwi-interacting) und die 4) long non-coding oder lncRNA 23.
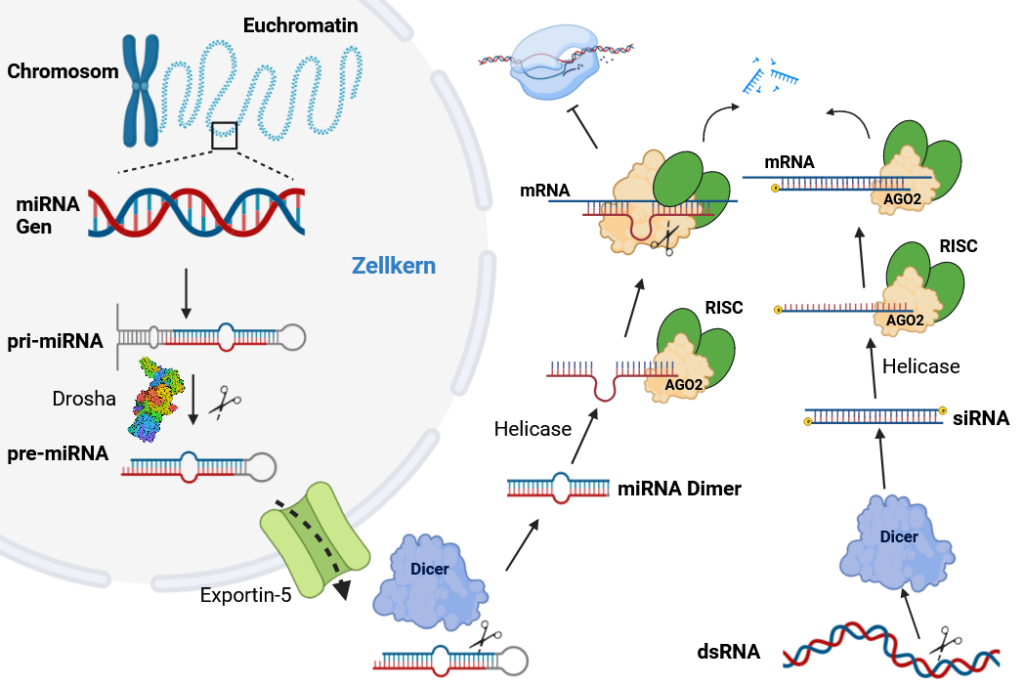
Die Wirkung von miRNA und siRNA ist fast identisch: beide umfassen 19-24 Nukleotide und bilden zusammen mit Argonauten-Proteinen einen RISC-Komplex (RNA induced silencing complex) aus (Abbildung 4) 23.
Der Unterschied zwischen ihnen ist ihre Herkunft, siRNAs werden aus langen doppelsträngigen RNAs geschnitten, während miRNAs aus kürzeren, einzelsträngigen RNAs stammen. Beide Vorstufen werden von dem passend benannten Dicer Enzym (engl. würfeln, Vgl. mit dem Küchenhelfer Nicer Dicer) zurechtgeschnitten.
Ihre Sequenz ist einigermaßen komplementär zu der von mRNAs. Dabei kann eine einzige miRNA hunderte von Genen regulieren, da nur 7-8 Nukleotide perfekt binden müssen (Seed Region) 25. Einmal gebunden, verhindern sie so die Translation zu Proteinen oder bewirken mit Hilfe des Argonautenproteins AGO2 direkt den Abbau der mRNA an Ort und Stelle. Von siRNAs ist ebenfalls bekannt, dass sie Stummschaltung durch Förderung von DNA- oder Histonmethylierung bewirken können 1.
Auch piRNAs sind unter 30 Nukleotid lange, einzelsträngige RNAs und primär in Keimzellen wichtig. Dort methylieren sie springende Genelemente (Transposons) 26, welche ansonsten wichtige Gene ausschalten könnten.
Die über 200 und bis zu 100 kb (kilobasen, also tausend) Nukleotid langen lncRNAs verbleiben meist im Zellkern, wo sie hauptsächlich mit Chromatin interagieren. Am besten erforscht ist bisher die X-inactive specific transcript (Xist) RNA, welches sich über das inaktivierte X-Chromosom legt, und somit Gene stilllegt 27.
Die Grenzen der ncRNA Mechanismen sind fließend, denn die besonders langen ncRNAs können auch als Blaupause für kleinere miRNAs 28 oder siRNAs 29 dienen.
Generationsübergreifende Vererbung – auch beim Menschen?
Wie wir schon gelernt haben, sind Körper- oder auch Somazellen in der Lage auf Signale aus der Umwelt zu reagieren, und ihr verändertes Programm an Tochterzellen weiterzugeben.
Nur Keimzellen, also Spermien- oder Eizellen, übertragen Erbinformationen an Nachkommen. In diesen werden epigenetische Marker jedoch gleich zweimal entfernt. Einmal 4-6 h nach der Befruchtung der Eizelle, wenn das Erbgut des Vaters (paternales Genom) fast komplett gesäubert wird von Methylierungen, während die maternale DNA nahezu unberührt bleibt 30. Die zweite, umfangreichere Säuberungswelle findet während der Ausbildung von ersten Vorläufer-Keimzellen im Embryo statt 30. Dabei wird auch die Chromatinstruktur auf den Kopf gestellt.
Bei Pflanzen hingehen entstehen Keimzellen häufig aus Somazellen, die bereits Umwelteinflüssen ausgesetzt waren. Viele Pflanzenarten können sich sogar klonal fortpflanzen, so dass Keimzellen hierfür gar nicht nötig sind 31. Somit ist es wenig überraschend, dass generationsübergreifende epigenetische Vererbung in Pflanzen häufig vorkommt.
Da der Fötus schon im Mutterleib Signale vom Stoffwechsel der Mutter oder anderen Umweltreizen erhält, ist es sehr schwierig, epigenetische Vererbung von generationsübergreifender Vererbung zu trennen. Epigenetische Marker müssen also mindestens noch in der dritten Generation zu beobachten sein, um von generationsübergreifender epigenetischer Vererbung zu reden 32.
Bei Mäusen kann man den mütterlichen Einfluss auf den Fötus umgehen, indem man Eizellen und Spermien im Reagenzglas (in vitro) mischt, zum Beispiel von übergewichtigen Mauseltern 33. Eine gesunde Leihmutter trägt dann die befruchtete Eizelle aus, um Einflüsse des gestörten Stoffwechsels der biologischen Mutter.
Ein bekanntes Experiment zum Nachweis generationsübergreifender Vererbung in Mäusen setzte die Eltern (F0) einem starken Geruchsstress aus (Acetophenon) 34. Roch sowohl die erste (F1), als auch die zweite Generation (F2) an Nachkommen diesen Stoff, waren sie ähnlich gestresst wie ihre Eltern. Man konnte zeigen, dass der verantwortliche Geruchsrezeptor Olfr151 aufgrund von unzureichender Methylierung in allen Tieren überrepräsentiert war. Die epigenetischen Marker wurden dabei durch die Keimzellen vererbt.
Menschen kann man nicht nach Belieben miteinander paaren, wodurch es sich sehr viel schwieriger gestaltet, generationsübergreifende Vererbung nachzuweisen.
Traumata und epigenetische Auswirkungen auf Kinder
Was sich sehr wohl nachweisen lässt, sind epigenetische Veränderungen des Embryos im Mutterleib. Als die Beispielstudie schlechthin gilt hierfür die niederländische Hungersnot 1944/1945 gegen Ende des zweiten Weltkriegs 35.
Eine Untersuchung der betroffenen Kinder im Vergleich mit ihren später geborenen Geschwistern zeigte Erschreckendes. So waren die Menschen, welche der pränatalen Mangelernährung ausgesetzt waren mit höherer Wahrscheinlichkeit übergewichtig, weniger stressresistent, arbeitslos (!), schnitten schlechter bei IQ-Tests ab und hatten ein erhöhtes Risiko für psychische Krankheiten wie Schizophrenie oder Depression. Selbst deren Nachkommen, also die Enkel, hatten mit Gewichtsproblemen zu kämpfen- sofern ihre Väter betroffen waren. Doch vermutet man hier eher eine Beeinflussung der sich bildenden Keimzellen in den Embryos als generationenübergreifende Vererbung.
Postnatale Traumata zeichnen sich ebenfalls in den Genen ab. Opfer von Kindesmissbrauch besitzen weniger Glucocorticoid-Rezeptoren in ihrem Hypocampus aufgrund erhöhter Methylierung 36. Dadurch können sie schlechter mit Stress umgehen und besitzen ein erhöhtes Suizidrisiko 37.
Gedächtnis wäre ohne das Epigenom nicht möglich
Doch auch positivere Dinge wie Lernen oder das Langzeitgedächtnis gründen auf epigenetischen Vorgängen 38. Dabei werden besonders immediate early genes (IEGs) als Schnellantwort auf Stimulus hochreguliert. Ein ausgeklügeltes System aus Demethylierung (Aktivierung) mehrerer hundert Gene und anschließender Neubildung von DNA-Methylierungen wird in Kraft gesetzt.
Für diesen Zweck bietet sich der anpassungsfähige und vor allem andauernde epigenetische Code geradezu an. Langanhaltende plastische Veränderungen des Gehirns, wie die Ausbildung von Erinnerungen, basieren auf dauerhaften funktionellen Anpassungen von Neuronen, keinen zeitlich begrenzten elektrischen Signalen.
Die molekulare Uhr – genetische Anzeichen für Alterung
Unsere Epigenetik ist nicht statisch, sondern ständig im Wandel. Bei einem Neugeborenen ist noch viel mehr DNA methyliert als bei einem 103-Jährigen. Ein 26-jähriges Genome liegt ungefähr dazwischen 39. Somit ist die Inaktivierung unnötiger Gene durchaus als positiv zu bewerten!
Das Altern kann aus biologischer Sicht als die fortschreitende Verkürzung der Chromosomenenden, den Telomeren, beschrieben werden. Dafür verantwortlich ist das Enzym Telomerase, welches durch starke psychische Belastung geschwächt werden kann. Dies äußert sich in einem beschleunigten Alterungsprozess. Für diese Entdeckung gab es 2009 den Medizin-Nobelpreis für Elizabeth Blackburn.
Aber auch epigenetische Anzeichen lassen sich nachweisen. So gibt es 353 CpG Stellen, deren Methylierungszustand Auskunft darüber geben, wie alt ein Mensch genetisch gesehen ist. Interessanterweise korrelieren eine gesunde Ernährung aus Fisch, Gemüse und Obst, sowie ein geringer BMI (body mass index) und ein hoher Bildungsgrad mit einem geringeren genetischen Alter. Ein erhöhter BMI bewirkt indes eine Beschleunigung des Alterungsprozesses 40. Somit spiegelt sich die Lebensweise auch im Genom wider.
Viele dieser 353 CpG Stellen befinden sich in der Nähe von bivalenten Genen oder aktiven Promotoren (angeschalteten Genen). Daher wird vermutet, dass das Altern mit einer geringeren Flexibilität bzw. Plastizität einhergeht. Also, dass der Körper nicht mehr so schnell zwischen zwei Genaktivitätszuständen hin- und herwechseln kann.
Verhalten und Umwelt verändern die Gene
Eine Studie zu epigenetischen Auswirkungen des Rauchens untersuchte 90 CpG Methylierungsmuster von insgesamt 4900 Lang-, Kurz- und Nicht-Rauchern 41. Dabei stellten sie fest, dass die epigenetischen Markierungen des Rauchens schnell verschwanden, wenn die Teilnehmer mit dem Qualmen aufhörten. In Abhängigkeit davon, wie lange und intensiv die Teilnehmer im Vorfeld rauchten, dauerte es 1 bis 2 Jahre, bis sie epigenetisch wieder als Nichtraucher eingeordnet wurden.
Auch moderate Feinstaub-Belastungen machen sich epigenetisch bemerkbar. Eine Studie des Umweltbundesamtes, durchgeführt in Stuttgart, konnte spezifische Methylierungsmuster als Antwort auf Feinstaubbelastung in 60 Teilnehmern zeigen. Auch bei der Häufigkeit von miRNAs gab es Unterschiede in den Atemwegen. Ob die Beobachtungen Biomarker für Erkrankungen sind, bleibt noch zu klären, da alle Probanden jung und ohne Vorerkrankung waren.
Abschließend lässt sich sagen, dass sowohl die eigene Lebensweise, als auch äußere Umstände, einen selbst oder die Eltern betreffend, nachhaltige Spuren im Genom hinterlassen. Das unterstreicht auch aus biologischer Sicht die Verantwortung für sich und die Umwelt.
Aus medizinischer Sicht ist die Epigenetik ein höchstspannendes Feld. Besonders bei Krebs sind therapeutische Ansätze bereits vielversprechend 42. Weitere Forschung wird Diagnosemöglichkeiten vielerlei Krankheiten aufgrund von Blutproben (und den epigenetischen Markern) ermöglichen.
Fandest Du diesen Beitrag hilfreich? Feedback und/oder Deine Meinung zum Thema als Kommentar oder per Kontaktformular sind immer willkommen.
Lust auf mehr? Weitere Artikel in diesem Stil findest Du hier.
Weiterführende Links
- Epigenetische Auswirkungen von Feinstaub: https://www.umweltbundesamt.de/publikationen/epigenetische-veraenderungen-unterschiedlicher
- Generationsübergreifende Vererbung bei übergewichtigen Mäusen: https://www.gesundheitsforschung-bmbf.de/de/epigenetik-essgewohnheiten-schlagen-sich-im-erbgut-nieder-3319.php
- Thermenbereich Epigenetik bei Spektrum der Wissenschaft: https://www.spektrum.de/thema/epigenetik/1191602
- https://www.spektrum.de/news/vaters-erbsuende/1258600
- Was ist Epigenetik von Peter Spork, dem Herausgeber des Newsletters für Epigenetik: https://www.riffreporter.de/de/wissen/was-ist-epigenetik
- Newsletter: https://www.newsletter-epigenetik.de/
- Essgewohnheiten und Epigenetik: https://www.gesundheitsforschung-bmbf.de/de/epigenetik-essgewohnheiten-schlagen-sich-im-erbgut-nieder-3319.php
Weitere empfehlenswerte Artikel:
- https://www.cdc.gov/genomics/disease/epigenetics.htm (englisch)
- https://www.planet-wissen.de/natur/forschung/epigenetik/index.html
- https://www.max-wissen.de/max-hefte/biomax-23-epigenetik/
- https://www.wissensschau.de/genom/epigenetik_und_epigenom.php
Quellen
1. Cavalli, G. & Heard, E. Advances in epigenetics link genetics to the environment and disease. Nature 571, 489–499 (2019).
2. Deans, C. & Maggert, K. A. What Do You Mean, “Epigenetic”? Genetics 199, 887–896 (2015).
3. Haig, D. Commentary: The epidemiology of epigenetics. Int. J. Epidemiol. 41, 13–16 (2012).
4. Ouni, M. & Schürmann, A. Epigenetic contribution to obesity. Mamm. Genome 31, 134–145 (2020).
5. Ahmed, F. Epigenetics: Tales of adversity. Nat. 2010 4687327 468, S20–S20 (2010).
6. Zhang, T., Cooper, S. & Brockdorff, N. The interplay of histone modifications – writers that read. EMBO Rep. 16, 1467–1481 (2015).
7. Ptashne, M. Epigenetics: Core misconcept. Proc. Natl. Acad. Sci. 110, 7101–7103 (2013).
8. Bonasio, R., Tu, S. & Reinberg, D. Molecular Signals of Epigenetic Marks. Science (80-. ). 330, 612–616 (2010).
9. Machida, S. et al. Structural Basis of Heterochromatin Formation by Human HP1. Mol. Cell 69, 385-397.e8 (2018).
10. Müller, M. M., Fierz, B., Bittova, L., Liszczak, G. & Muir, T. W. A two-state activation mechanism controls the histone methyltransferase Suv39h1. 12, 188–193 (2016).
11. Ginno, P. A., Burger, L., Seebacher, J., Iesmantavicius, V. & Schübeler, D. Cell cycle-resolved chromatin proteomics reveals the extent of mitotic preservation of the genomic regulatory landscape. Nat. Commun. (2018). doi:10.1038/s41467-018-06007-5
12. Teves, S. S. et al. A stable mode of bookmarking by TBP recruits RNA polymerase II to mitotic chromosomes. 1–22 (2018).
13. Inoue, A., Jiang, L., Lu, F., Suzuki, T. & Zhang, Y. Maternal H3K27me3 controls DNA methylation-independent imprinting. Nature 547, 419–424 (2017).
14. Miska, E. A. & Ferguson-Smith, A. C. Transgenerational inheritance: Models and mechanisms of non-DNA sequence-based inheritance. Science 354, 59–63 (2016).
15. Teif, V. B. et al. Nucleosome repositioning links DNA (de)methylation and differential CTCF binding during stem cell development. Genome Res. 24, 1285 (2014).
16. Greenberg, M. V. C. & Bourc’his, D. The diverse roles of DNA methylation in mammalian development and disease. Nat. Rev. Mol. Cell Biol. 20, 590–607 (2019).
17. Li, E. & Zhang, Y. DNA methylation in mammals. Cold Spring Harb. Perspect. Biol. 6, (2014).
18. Okano, M., Bell, D. W., Haber, D. A. & Li, E. DNA methyltransferases Dnmt3a and Dnmt3b are essential for de novo methylation and mammalian development. Cell 99, 247–257 (1999).
19. Tucci, V. et al. Genomic Imprinting and Physiological Processes in Mammals. Cell 176, 952–965 (2019).
20. Tang, W. W. C. et al. A Unique Gene Regulatory Network Resets the Human Germline Epigenome for Development. Cell 161, 1453–1467 (2015).
21. Lyko, F. et al. The Honey Bee Epigenomes: Differential Methylation of Brain DNA in Queens and Workers. PLoS Biol. 8, e1000506 (2010).
22. Chittka, A. & Chittka, L. Epigenetics of Royalty. PLoS Biol. 8, (2010).
23. Wei, J.-W., Huang, K., Yang, C. & Kang, C.-S. Non-coding RNAs as regulators in epigenetics. Oncol. Rep. 37, 3–9 (2017).
24. Dunham, I. et al. An integrated encyclopedia of DNA elements in the human genome. Nat. 2012 4897414 489, 57–74 (2012).
25. Lewis, B. P., Burge, C. B. & Bartel, D. P. Conserved seed pairing, often flanked by adenosines, indicates that thousands of human genes are microRNA targets. Cell 120, 15–20 (2005).
26. Kuramochi-Miyagawa, S. et al. DNA methylation of retrotransposon genes is regulated by Piwi family members MILI and MIWI2 in murine fetal testes. Genes Dev. 22, 908–917 (2008).
27. Clemson, C. M., McNeil, J. A., Willard, H. F. & Lawrence, J. B. XIST RNA paints the inactive X chromosome at interphase: evidence for a novel RNA involved in nuclear/chromosome structure. J. Cell Biol. 132, 259–275 (1996).
28. Cai, X. & Cullen, B. R. The imprinted H19 noncoding RNA is a primary microRNA precursor. RNA 13, 313 (2007).
29. Ogawa, Y., Sun, B. K. & Lee, J. T. Intersection of the RNA interference and X-inactivation pathways. Science 320, 1336–1341 (2008).
30. Hajkova, P. Epigenetic reprogramming in the germline: towards the ground state of the epigenome. Philos. Trans. R. Soc. B Biol. Sci. 366, 2266 (2011).
31. Heard, E. & Martienssen, R. A. Transgenerational Epigenetic Inheritance: Myths and Mechanisms. Cell 157, 95–109 (2014).
32. Kaspar, D., Hastreiter, S., Irmler, M., Hrabé de Angelis, M. & Beckers, J. Nutrition and its role in epigenetic inheritance of obesity and diabetes across generations. Mamm. Genome 31, 119–133 (2020).
33. Huypens, P. et al. Epigenetic germline inheritance of diet-induced obesity and insulin resistance. Nat. Genet. 2016 485 48, 497–499 (2016).
34. Dias, B. G. & Ressler, K. J. Parental olfactory experience influences behavior and neural structure in subsequent generations. Nat. Neurosci. 17, 89–96 (2014).
35. Roseboom, T. J. Epidemiological evidence for the developmental origins of health and disease: effects of prenatal undernutrition in humans. J. Endocrinol. 242, T135–T144 (2019).
36. McGowan, P. O. et al. Epigenetic regulation of the glucocorticoid receptor in human brain associates with childhood abuse. Nat. Neurosci. 12, 342–348 (2009).
37. Yehuda, R. & Lehrner, A. Intergenerational transmission of trauma effects: putative role of epigenetic mechanisms. World Psychiatry 17, 243–257 (2018).
38. Bernstein, C. DNA Methylation and Establishing Memory. Epigenetics Insights 15, 251686572110724 (2022).
39. Heyn, H. et al. Distinct DNA methylomes of newborns and centenarians. Proc. Natl. Acad. Sci. 109, 10522–10527 (2012).
40. Quach, A. et al. Epigenetic clock analysis of diet, exercise, education, and lifestyle factors. Aging (Albany. NY). 9, 419–446 (2017).
41. McCartney, D. L. et al. Epigenetic signatures of starting and stopping smoking. EBioMedicine 37, 214–220 (2018).
42. Ahuja, N., Sharma, A. R. & Baylin, S. B. Epigenetic Therapeutics: A New Weapon in the War Against Cancer. Annu. Rev. Med. 67, 73 (2016).

